Gentechnische Methoden
Gentechnische Verfahren
1. PCR – Polymerase-Kettenreaktion
Die PCR (engl. Polymerase Chain Reaction) ist eine Methode, um die DNA zu vervielfältigen, ohne einen lebenden Organismus, wie z.B. Escherichia coli oder Hefe zu benutzen.
Dieses Verfahren hat man sich in der Natur abgeschaut. Sie ist der natürlichen Replikation sehr ähnlich und erfolgt in 4 Schritten:
- SchmelzenMan nimmt ein doppelsträngiges DNA - Molekül und erhitzt es auf 96°C. Nun trennen sich die 2 Stränge voneinander, da die Wasserstoffbrückenbindungen gelöst werden. Dann gibt man kurze synthetische DNA - Fragmente hinzu, die Primer. Diese sind komplementär zu den Enden der DNA.
- HybridisierungDie Primer setzten sich beim Abkühlen auf 68°C an die DNA-Einzelstränge. Es bilden sich also kurze Doppelstrangabschnitte. Diese dienen als Startpunkte für die Polymerase und verhindern, dass sich die 2 Stränge wieder zusammensetzen.
- ReplikationDas Gemisch wird nun auf 72°C erhitzt. Die Polymerase beginnt zu arbeiten und repliziert die Stränge.Nach Beendigung sind aus 2 Einzelsträngen 2 Doppelstränge geworden, die identisch sind.
- ZykluswiederholungDieser Doppelstrang wird durch Erhöhung der Temperatur wieder in 2 Einzelstränge getrennt. Nun sind die Bindungsstellen für die Primer erneut frei und der Vorgang kann nach dem Absinken der Temperatur von neuem starten. Dies wird mehrmals wiederholt. Nach ca. 20 Zyklen hat man eine millionenfache Anreicherung des zwischen den Primern liegenden DNA – Abschnitts.
Man verwendet Polymerasen aus thermophilen (hitzeliebenden) Bakterien, da „normale“ Polymerasen bei der Erhitzung auf 96°C kaputt gehen.
Dies vereinfachte den Prozess, da man nicht immer neue Polymerase dazu geben musste. Diese Methode findet „in vitro“, also im Reagenzglas statt. Die natürliche Form nennt man „in vivo“.
Dies ist eine exponentielle Vervielfältigung des DNA - Abschnittes = Amplifizierung. Die entstandenen DNA Abschnitte = Amplifikate. Sie ist ähnlich der natürlichen Replikation.
Dies wird z. B. angewandt:
- in der Kriminalistik (gen. Fingerabdruck) oder beim Vaterschaftstest
- in der medizinischen Diagnostik, wenn im Blut Viren-DNA nachzuweisen ist
- in der Evolutionsbiologie, um Verwandtschaftsbeziehungen und Abstammungslinien zu verfolgen
- zur Erkennung von Erbkrankheiten
- zur Klonierung von Genen
- bei Mutagenese (Veränderung der DNA-Sequenz)
2. Sequenzanalyse(= Sequenzierung)
Dies ist eine Methode zur Entschlüsselung der Basensequenz der Erbinformation.Heutzutage ist das meist ein automatisierter und computergestützter Prozess in der Molekularbiologie und der Bioinformatik.
Die Durchführung erfolgt meist nach der Methode von Sanger:
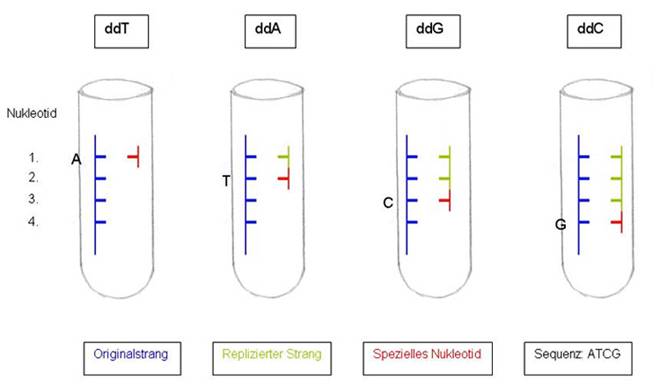
Man hat 4 Reagenzgläser. In jedes gibt man die 4 unterschiedlichen Nukleotide, die einsträngige Original-DNA und jeweils ein spezielles Nukleotid mit der jeweiligen Base A T G C. Dieses spezielle Nukleotid heißt: ddA, ddT, ddC, ddG. Das dd steht für Didesoxyribonukleotid und hat die Eigenschaft die Synthese des Gegenstranges durch die DNA-Polymerase abzubrechen. Die Replikation des Gegenstranges läuft also so lange, bis sich dieses spezielle Nukleotid ansetzt. Dadurch entstehen unterschiedlich lange DNA-Stränge. Wenn man weiß, dass sich am Ende des Stranges dieses Nukleotid befindet, kennt man somit auch das der Original – DNA. Nun vergleicht man die unterschiedlich langen DNA Stränge (mittels Elektrophorese im Agarose-Gel ) und notiert sich die Basen auf dem Originalstrang. Somit hat man die Originalsequenz der DNA.
DNA-Auftrennung
Beispiel:
Im Röhrchen mit dem speziellen Nukleotid ddT hat sich dieses an der ersten Stelle angelagert. Somit befindet sich auf dem Originalstrang das Nukleotid A. Im zweiten Röhrchen hat sich ddA am zweiten Nuleotid angelagert, d.h. auf dem Originalstrang ist das Nukleotid T. Ebenso funktioniert das in den anderen 2 Röhrchen. So erhält man die Sequenz: ATCG
Dieses Verfahren nutz man in erster Linie zur Bestimmung der Basenabfolge von bestimmten charakteristischen Abschnitten, den Genen.
In Datenbanken werden bereits entschlüsselte Sequenzen archiviert. Dadurch ist man in der Lage, wichtige Aufschlüsse über Funktion unbekannter Gene zu gewinnen. Man kann die neuen unbekannten mit den bereits gesicherten Sequenzen vergleichen. Somit lässt sich häufig die Bedeutung oder Funktion unerforschter Gene vorhersagen.
3. Blotting
Blotting ist eine molekularbiologische Arbeitstechnik, bei der Nucleinsäuren (DNA), Ribonucleinsäuren (RNA) oder Proteine auf Folienmaterial übertragen und dort fixiert werden.
"Southern" Blotting wurde von Edwin Southern entwickelt und 1975 veröffentlicht. Seine Methode bezog sich auf DNA-Sequenzen und wurde weiterentwickelt zum Northern-Blot (RNA-Sequenzen) und Western-Blot (Proteine).
Die Methode am Beispiel vom Western-Blot:
- Vorbereitung der Proben: Die Proben werden mit SDS (Sodiumdodecylsulfat) und einem Reduktionsmittel bei 70°C inkubiert. Ergebnis ist die vollständige Denaturierung der Proteine. Außerdem besitzen alle Moleküle durch das SDS nun die gleiche negative Ladung und werden somit nach ihrer Größe aufgetrennt, ohne Berücksichtigung ihrer ursprünglichen Ladung.
- Gelelektrophorese: Für die Gelelektrophorese benötigt man ein Gradientengel aus Polyacrylamid. Es besteht aus Sammel- und Trenngel. Das Sammelgel besitzt gleich große Poren, wo die Proben aufgetragen werden und es somit eine einheitliche Startlinie gibt. Durch Anlegen einer Spannung wandern die negativ geladenen Proteine nun von der Kathode (- Pol) zur Anode (+ Pol) und passieren dabei das Trenngel. Dieses ist netzartig aufgebaut und die Porengröße nimmt in Richtung Anode ab. Kleinere Moleküle können dadurch weiter wandern, als die Größeren. Die Probe trennt sich somit auf.Polyacrylamid-Gel mit aufgetrennten Proteinen, angefärbt mit Coomassie-Färbelösung:
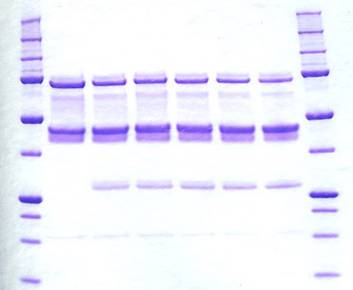
- Blotting Auf die Anode der Blotkammer wird ein Filterpapier gelegt und darauf die vorbereitete PVDF-Membran. Nun folgt das Gel und anschließend wieder ein Filterpapier. Der Deckel der Blotkammer mit der Anode wird kurz aufgedrückt und eine Spannung angelegt. Die Moleküle wandern nun (von der Kathode Richtung Anode) aus dem Gel auf die Membran und binden dort. Anschließend wird diese in 5% Magermilchpulver gelegt, um die noch freien Bindungsstellen auf ihr zu besetzen. Es folgen mehrere Waschschritte und anschließend die Zugabe von Antikörpern, die sich spezifisch (nach dem Schlüssel-Schloss-Prinzip) an die gesuchten Proteine heften. Nach weiteren Waschschritten können die angehefteten Antikörper durch eine Farbreaktion nachgewiesen werden.
Anwendungsgebiete:
Western-Blot:
- in der Gentechnik, nach Transformation an Zellen und anschließendem Nachweis von bestimmten Proteinen
Southern-Blot:
- Überprüfung, wie viele Kopien eines Gens im Erbgut vorhanden sind
- Nachweis, ob sich Gene, die im Organismus A enthalten sind, auch im Organismus B befinden
Northern-Blot:
- zur Untersuchung, welche Proteine (entsprechende RNA-Suche) in einer Zelle exprimiert werden
Wird ein Gen in verschiedenen Geweben bzw. zu verschiedenen Zeitpunkten in der Entwicklung aktiv? (mRNA wird aus dem Gewebe bzw. zum fraglichen Zeitpunkt extrahiert.)
Bei den verschiedenen Ausgangsproben werden jeweils entsprechende Gele bei der Elektrophorese, sowie entsprechende Membranen beim Blotten benutzt. Neben dem beschriebenen Elektroblotting gibt es zum Beispiel noch das Kapillarblotting, wo sich die Kapillarkräfte von Filterpapieren zunutze gemacht werden, oder das Vakuum- oder Überdruckblotting.
Quellenangaben
http://www.biosicherheit.de/lexikon/
http://de.wikipedia.org/
http://www.lgl.bayern.de/de/left/fachinformationen/lebensmittel/gentechnik/gentechnik-ix.htm
http://www.gesundheit.de/roche/ro07500/r8264.html
http://www.academicus.ch/de/dna_sequenzanalyse.html
http://www.schulserver.hessen.de/frankfurt/paul-ehrlich/blotten/blotting.htm
Bundesministerium für Bildung und Forschung – “Biotechnologie – Basis für Innovationen”